Retour
Galerie des structures

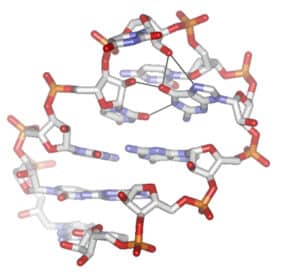
PDB entries: 2FCY, 2FCZ, 2FD0, 2FCX
References: Ennifar E, Paillart JC, Bodlenner A, Walter P, Weibel JM, Aubertin AM, Pale P, Dumas P & Marquet R, Nucleic Acids Res (2006) Ennifar E, Paillart JC, Bernacchi S, Walter P, Pale P, Decout JL, Marquet R & Dumas P, Biochimie (2006)

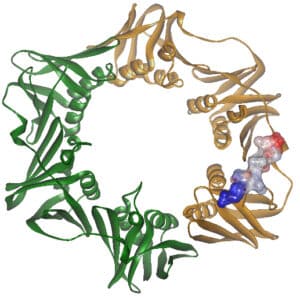
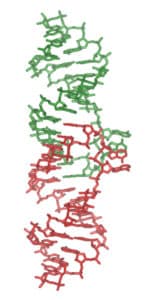
PDB entries: 2QEK, 3C3Z, 3C44, 3C5D, 3C7R
Reference: Freisz S, Lang K, Micura R, Dumas P & Ennifar E, Angewandte Chemie (2008)
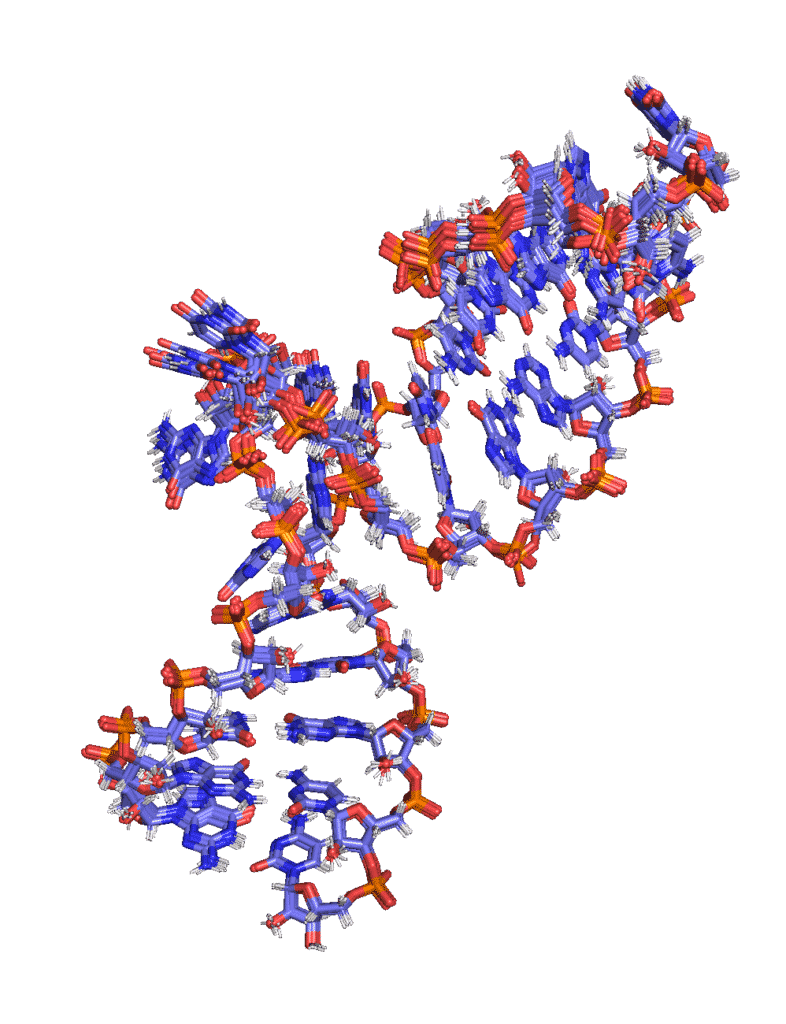
PDB entrie: 6HYK
Reference : M.E. Chagot, M. Quinternet, B. Rothé, B. Charpentier, J. Coutant, X. Manival* & I. Lebars*, 2019, “The yeast C/D box snoRNA U14 adopts a “weak” K-turn like conformation recognized by the Snu13 core protein in solution”, Biochimie, 2019, 164: 70-82.
 Intranet
Intranet Acces
Acces Contact
Contact