Back
Structure gallery

Genomic RNA
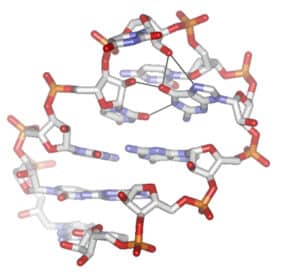
Dimerization Initiation Site kissing-complex bound to aminoglycoside antibiotics neamine, ribostamycin, neomycin and lividomycin
PDB entries: 2FCY, 2FCZ, 2FD0, 2FCX References: Ennifar E, Paillart JC, Bodlenner A, Walter P, Weibel JM, Aubertin AM, Pale P, Dumas P & Marquet R, Nucleic Acids Res (2006) Ennifar E, Paillart JC, Bernacchi S, Walter P, Pale P, Decout JL, Marquet R & Dumas P, Biochimie (2006)

Genomic RNA
Dimerization Initiation Site
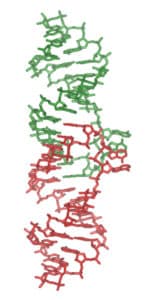
extended duplex unliganded and bound to aminoglycoside antibiotics ribostamycin, paromomycin, neomycin and lividomycin
PDB entries: 2QEK, 3C3Z, 3C44, 3C5D, 3C7R Reference: Freisz S, Lang K, Micura R, Dumas P & Ennifar E, Angewandte Chemie (2008)
 Intranet
Intranet Access
Access Contact
Contact