Notre objectif est de comprendre le mécanisme moléculaire des machines biomoléculaires en utilisant une approche biophysique intégrée, combinant la biochimie, la thermodynamique, la cinétique, la biologie structurale et la dynamique moléculaire.

Axes de recherche
Contacts : Eric Ennifar, Isabelle Lebars.
Collaborateurs : Joao Marques (Federal University of Minas Gerais, Brazil & CNRS UPR 9022 IBMC), Karim Majzoub (IGMM Montpellier, CNRS UMR 5535).
Les virus du Zika (ZIKV) et de la Dengue (DENV) appartenant à la famille des Flavivirus sont transmis par piqûres de moustiques infectés. Avec environ 390 millions d’infections répertoriées chaque année, DENV est considéré comme l’un des virus les plus critiques par l’Organisation Mondiale de la santé. Concernant le ZIKV, la première émergence significative a été observée en 2007 dans les îles du Pacifique Sud et plus tard au Brésil, se répandant rapidement sur le continent Américain en 2015-2016. Actuellement, aucun traitement efficace ou vaccin n’existent contre ces virus. Dans la mesure où DENV et ZIKV se répandent chez des hôtes vertébrés et invertébrés, une stratégie efficace pour bloquer la transmission virale et la diffusion du virus serait de cibler des étapes spécifiques de la réplication virale. Le développement de nouvelles stratégies antivirales nécessite une compréhension précise des étapes clefs du cycle viral. Les virus à ARN comportent souvent, au sein de leur génome, des régions structurées non codantes multifonctionnelles. Les régions non traduites 5’- et 3’- (UTR) des génomes de DENV et ZIKV sont hautement conservées et sont impliquées dans la réplication virale.

Les interactions ARN-proteines jouent un rôle crucial dans ce cycle de réplication et dans le détournement de la machinerie cellulaire de l’hôte. Afin de développer de nouvelles stratégies antivirales, nous avons pour objectifs de : (i) décrypter les interactions ARN-protéines impliquées dans le cycle de réplication et (ii) identifier et caractériser les facteurs chez le moustique qui interagissent avec l’ARN génomique de ces virus. Dans ce but, nous utilisons des approches biophysiques intégrées incluant la microcalorimétrie isothermale (ITC), la technologie « switchSENSE » basée sur l’utilisation de puces à ADN, la spectrométrie UV-visible, la Spectrométrie de Masse (MS), la Résonance Paramagnétique Electronique (RPE, en collaboration), la Cryo-Microscopie Electronique (Cryo-EM), la Résonance Magnétique Nucléaire (RMN) et la Cristallographie aux rayons X.
A venir
Chez les eucaryotes, l’initiation de la traduction débute par le recrutement du ribosome sur l’ARNm à l’aide d’une ‘coiffe’ m7G en 5’ et des facteurs d’initiation (eIFs), suivis par la reconnaissance fiable de son codon d’initiation. Le recrutement du ribosome sur l’ARNm est suivi par l’assemblage ordonné des différents complexes d’initiation (CIs): i) 48S actif (48S CI) puis, suite à l’interaction avec la grande sous-unité ribosomique 60S, ii) 80S CI. Bien que prédominant pour les ARNm cellulaires, ce modèle classique d’initiation de la traduction est compromis dans des conditions de stress, notamment celles liées au microenvironnement des cellules tumorales, telles que l’hypoxie, la privation de nutriments et la réponse immunitaire. Néanmoins, la traduction d’un sous-ensemble d’ARNm cellulaires codant pour des régulateurs du développement tumoral est maintenue par des mécanismes alternatifs faisant appel à des protéines de liaison à l’ARN ou des éléments contenus dans l’ARNm lui-même (les sites d’entrée des ribosomes internes (IRES), modifications d’ARN, G-quadruplexes, µORF), souvent retrouvés dans le 5’UTR de leur mRNA. Parmi eux, l’ARNm du facteur 1α induit par l’hypoxie (HIF-1α) et de la β-caténine sont particulièrement intéressants car le maintien de leur traduction favorise la persistance et la plasticité des cellules cancéreuses. A ce jour, nos connaissances sur un éventuel mécanisme de traduction alternatif soutenant la traduction de l’ARNm de HIF-1α et β-caténine sous hypoxie doivent encore être prouvées et plusieurs questions restent ouvertes : i) quels sont les déterminants structurels et/ou de séquence pour leur traduction lorsque l’initiation canonique est altérée ? ii) Comment le ribosome interagit-il avec ces déterminants ? La structure de la coiffe M7G est-elle nécessaire ? Existe-t-il des eIFs spécifiques et/ou des trans-acting factors nécessaires ?

L’objectif principal de notre recherche est d’obtenir un aperçu au niveau atomique du mécanisme de l’initiation alternative de la traduction induite par l’hypoxie régulant l’expression de HIF-1α et de la β-caténine en utilisant une combinaison d’approches biochimiques (cartographie de la structure de l’ARN, traduction in vitro et transfection de l’ARN cellulaire), biophysiques (thermodynamique et cinétique) et de biologie structurale (Cristallographie aux rayons X et cryomicroscopie électronique, cryo-EM).
Cette étude devrait donc permettre d’identifier les caractéristiques structurales des ARNm et/ou les facteurs jouant un rôle central dans la traduction des ARNm HIF-1α et β-caténine pendant la privation d’O2, qui pourraient devenir des cibles pour une nouvelle classe de médicaments anticancéreux.

A venir
A venir

Projets passés
Contact : Isabelle LEBARS.
Collaboratrice: Dr Anne-Catherine Dock-Bregeon (LBI2M, Roscoff, CNRS UMR 8227).
Le petit ARN nucléaire 7SK (7SKsnRNA) joue un rôle majeur dans la régulation de l’activité de l’ARN polymérase II en séquestrant et en inhibant le facteur d’élongation P-TEFb dans le complexe ribonucléoprotéique 7SKsnRNP. Ce processus est médié par la protéine HEXIM. P-TEFb, hétérodimère composé de la cycline T1 et de la kinase CDK9, est un facteur cellulaire indispensable recruté par la protéine virale Tat afin d’assurer la réplication de l’ARN viral dans le cycle d’infection par le virus de l’immunodéficience humaine (VIH-1). En déplaçant la protéine HEXIM de la partie 5’ de l’ARN 7SK (Hairpin I, HPI), la protéine virale Tat capture P-TEFb, le libérant du complexe 7SKsnRNP et permettant ainsi l’activation de la transcription.

Cette tige-boucle HPI, comprenant le motif « signature » de l’ARN 7SK, a fait l’objet de 3 études structurales indépendantes visant à identifier les facteurs structuraux à l’origine de la reconnaissance par HEXIM et Tat. Ces deux protéines contiennent des motifs hautement similaires « ARM » (Arginine Rich Motif) de liaison à l’ARN. De manière intéressante, 4 structures distinctes ont été obtenues dans différentes conditions (pH, sels). Nous avons utilisé la Titration Calorimétrique Isotherme (ITC) afin de déterminer les signatures thermodynamiques de l’interaction des domaines ARM de Tat et HEXIM avec l’ARN 7SK, dans différentes conditions. Nous avons ainsi montré que, bien que Tat-ARM et HEXIM-ARM soient hautement similaires, leurs signatures thermodynamiques dans le cadre de la liaison à HP1 diffèrent, indiquant des mécanismes de liaison distincts.
Publication :
Brillet K, Martinez-Zapien D, Bec G, Ennifar E, Dock-Bregeon A C, Lebars I. Different views of the dynamic landscape covered by the 5′-hairpin of the 7SK small nuclear RNA. RNA (2020), 26 (9):1184-1197.https://pubmed.ncbi.nlm.nih.gov/32430362/
Financement :

La transcriptase inverse (RT) du VIH-1 est une enzyme clé dans le cycle de vie du virus, qui convertit l’ARN génomique viral en ADN proviral. Elle est également une pierre angulaire de la thérapie anti-VIH, puisque la moitié des composés individuels inhibant la réplication virale ciblent le site actif de la polymérase de la RT. Les inhibiteurs RT du VIH-1 sont divisés en inhibiteurs RT analogues aux nucléosides (INTI), qui entrent en concurrence avec le substrat nucléosidique naturel, et en inhibiteurs RT non nucléosidiques (INNTI), qui se lient à une poche hydrophobe adjacente au site actif de la polymérase. Nous avons effectué des études biophysiques et structurelles sur deux nouveaux inhibiteurs de la RT : un analogue de DABO qui est apparu comme l’un des inhibiteurs de la RT les plus actifs signalés jusqu’à présent (Ec50 = 25 pM), et DAVP-1, un inhibiteur de la RT non nucléotidique avec un mécanisme d’action inhabituel. La structure cristalline de la RT du VIH-1 liée au DAVP-1 a révélé un nouveau site de liaison proche du site catalytique de la RT polymérase.
Nous avons également abordé le mécanisme d’action de la RT et des inhibiteurs non nucléosidiques de la RT (INNTI) par calorimétrie à titrage isotherme (ITC). En utilisant une nouvelle approche de CTI incrémentielle, une dissection thermodynamique étape par étape de l’activité de polymérisation RT a montré que la majeure partie de la force motrice pour la synthèse de l’ADN est fournie par la liaison initiale du dNTP. De manière surprenante, les données thermodynamiques et cinétiques ont conduit à une réinterprétation du mécanisme d’inhibition des INNTI. La liaison des INNTI aux complexes RT/ADN préformés est entravée par une barrière cinétique et les INNTI interagissent principalement avec la RT libre. Une fois formés, les complexes RT/INNTI se lient à l’ADN soit dans une orientation apparemment compatible avec la polymérase, soit forment des complexes à haute affinité en cul-de-sac, les deux complexes RT/INNTI/ADN étant incapables de se lier au substrat nucléotidique entrant.



Publications sélectionnées :
- Bec G, Meyer B, Gerard MA, Steger J, Fauster K, Wolff P, Burnouf D, Micura R, Dumas P & Ennifar E (2013). Thermodynamics of HIV-1 Reverse Transcriptase in action elucidates the mechanism of action for non-nucleoside inhibitors. J Am Chem Soc, 135(26):9743-52.
- Freisz S, Bec G, Radi M, Wolff P, Crespan E, Angeli L, Dumas P, Maga G, Botta M & Ennifar E (2010). Crystal Structure of the HIV-1 Reverse Transcriptase Bound to a Non-Nucleoside Inhibitor with a Novel Mechanism of Action. Angew Chem Int Ed Engl, 49(10):180
Les riboswitches sont des éléments non codants en amont ou en aval des ARN messagers qui, en se liant à un ligand spécifique, régulent l’initiation de la transcription ou de la traduction dans les bactéries, ou l’épissage alternatif dans les plantes et les champignons. Nous avons étudié les domaines aptamères des riboswitches thiamine pyrophosphate (TPP) du thiC et du thiM chez E. coli, qui régulent respectivement la transcription et la traduction, et celui du THIC chez A. thaliana. Pour tous, nous avons établi un mécanisme d’ajustement induit avec une liaison initiale lâche du TPP (caractérisée par les paramètres kon et koff) transformée ensuite en liaison serrée après un changement de conformation de l’ARN (caractérisé par les paramètres kF et kU). En utilisant notre approche kinITC, nous avons obtenu la dépendance en température de tous les paramètres cinétiques et thermodynamiques pour les riboswitches bactériens. Les résultats impliquent une régulation cinétique, qui nécessite que l’ARN polymérase s’arrête après la synthèse de l’aptamère du riboswitch. Un modèle quantitatif de régulation a mis en évidence la façon dont le temps de pause doit être lié aux taux cinétiques de liaison initiale du TPP pour obtenir un interrupteur ON/OFF dans la plage de concentrations correcte du TPP. Nous avons vérifié l’existence de ces pauses et la prédiction du modèle sur leur durée. Le riboswitch d’A. thaliana, au contraire, est sous contrôle thermodynamique classique et a montré une dépendance considérable de sa cinétique de pliage par rapport à la température, ce qui suggère qu’il pourrait également agir comme un thermocapteur. Notre analyse a également montré que les riboswitches à régulation cinétique réagissent plus fortement aux variations de concentration de leur ligand que les riboswitches à régulation thermodynamique. Cela rationalise l’intérêt de la régulation cinétique

Publications sélectionnées :
- Guedich S., Puffer-Enders B., Baltzinger M., Hoffmann G., Da Veiga C., Jossinet F., Thore S., Bec G., Ennifar E., Burnouf D., Dumas P. (2016). Quantitative and predictive model of kinetic regulation by E. coli TPP riboswitches. RNA Biol, 13(4):373-90.
- Burnouf D.Y., Ennifar E., Guedich S., Puffer B., Hoffmann G., Bec G., Disdier F., Baltzinger M, Dumas P. (2012). kinITC : a new method for obtaining joint thermodynamic and kinetic data by Isothermal Titration Calorimetry. J Am Chem Soc, 134(1):559-565.
Le site d’initiation de la dimérisation du VIH-1 (DIS) est une épingle à cheveux conservée dans le 5′ UTR de l’ARN génomique. L’altération du DIS affecte la dimérisation de l’ARN, l’emballage et la transcription inverse, et réduit l’infectivité virale. La boucle DIS initie la dimérisation du génome en formant un complexe boucle-boucle et est ensuite stabilisée, vraisemblablement sous une forme duplex étendue par interaction avec la protéine nucléocapside NCp7 virale. Nous avons résolu les structures cristallines de la boucle à baiser DIS et du duplex étendu. Nos structures ont révélé des similitudes avec le site A de l’ARN ribosomique 16S de la bactérie, qui est la cible des antibiotiques aminoglycosidiques. En conséquence, nous avons montré que les aminoglycosides se lient au VIH-1 DIS. Étonnamment, l’affinité de ces molécules pour la boucle à baiser du DIS est plus élevée que pour leur cible naturelle dans les ribosomes bactériens. Leur liaison stabilise fortement le complexe boucle-boucle et empêche sa conversion en forme duplex, même en présence de NCp7. Nous avons également résolu de nombreuses structures radiologiques à haute résolution du complexe DIS kissing-loop et du duplex étendu lié à plusieurs aminoglycosides. Cependant, l’identification des forces motrices moléculaires importantes pour la liaison DIS/aminoglycoside n’a pas pu être réalisée en considérant uniquement les données structurelles, ce qui souligne l’importance de la collecte de données thermodynamiques et structurelles pour une compréhension complète du processus de reconnaissance moléculaire.
En utilisant la microcalorimétrie ITC, la thermodynamique des aminoglycosides se liant aux formes duplex et kissing-loop du VIH-1 DIS a été étudiée dans une série de conditions. Avec les données structurelles fournies par les structures cristallines à haute résolution, nous avons pu établir les bases de la spécificité de la reconnaissance des médicaments/ARN et permettre de discriminer entre des sites de liaison spécifiques et des sites secondaires concurrents potentiels. Nous avons également pu montrer que la liaison du médicament au complexe DIS kissing-loop inhibe la conversion assistée par NCp7 dans la forme duplex étendue. En utilisant toutes ces données, nous pourrions rationnellement concevoir un conjugué aminoglycoside qui se lie spécifiquement à l’ARN DIS du VIH-1. Nos résultats montrent qu’il est possible de cibler le dimère du VIH-1 DIS avant et après la maturation de l’ARN assistée par le NCp7 avec la même molécule.


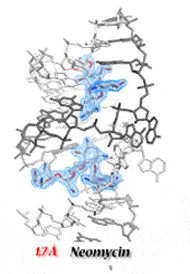
Publications sélectionnées :
- Ennifar E, Walter P, Ehresmann B, Ehresmann C, Dumas P (2001). Crystal structures of coaxially stacked kissing complexes of the HIV-1 RNA dimerization initiation site. Nat Struct Biol, 8(12):1064-8.
- Ennifar E, Paillart JC, Marquet R, Ehresmann B, Ehresmann C, Dumas P, Walter P (2003). HIV-1 RNA dimerization initiation site is structurally similar to the ribosomal A site and binds aminoglycoside antibiotics. J Biol Chem, 278(4):2723-30.
- Bernacchi S, Ennifar E, Tóth K, Walter P, Langowski J, Dumas P (2005). Mechanism of hairpin-duplex conversion for the HIV-1 dimerization initiation site. J Biol Chem, 280(48):40112-21.
- Ennifar E, Paillart JC, Bodlenner A, Walter P, Weibel JM, Aubertin AM, Pale P, Dumas P, Marquet R (2006). Targeting the dimerization initiation site of HIV-1 RNA with aminoglycosides : from crystal to cell. Nucleic Acids Res, 34(8):2328-39.
- Bernacchi S, Freisz S, Maechling C, Spiess B, Marquet R, Dumas P, Ennifar E (2007).Aminoglycoside binding to the HIV-1 RNA dimerization initiation site : thermodynamics and effect on the kissing-loop to duplex conversion. Nucleic Acids Res, 35(21):7128-39.
- Freisz S, Lang K, Micura R, Dumas P, Ennifar E (2008). Binding of aminoglycoside antibiotics to the duplex form of the HIV-1 genomic RNA dimerization initiation site. Angew Chem Int Ed Engl, 47(22):4110-3.
- Ennifar E, Aslam MW, Strasser P, Hoffmann G, Dumas P, van Delft FL (2013). Structure-guided discovery of a novel aminoglycoside conjugate targeting HIV-1 RNA viral genome. ACS Chem Biol, 8(11):2509-17.
à compléter
Publications sélectionnées :
- Freisz S., Mezher J., Hafirassou L., Wolff P., Nomine Y., Romier C., Dumas P., Ennifar E. (2012). Sequence and structure requirements for specific recognition of HIV-1 TAR and DIS RNA by the HIV-1 Vif protein. RNA Biol, 9(7):966 – 977.
Nous avons résolu une structure d’ARN 27-nt à une résolution subatomique (0,57 Å). À cette résolution, la plupart des atomes d’hydrogène de l’ARN, ainsi que certains hydrogènes du solvant aqueux, sont visibles. En outre, à une telle résolution ultime, le modèle d’atomes sphériques indépendants ne peut pas être appliqué et les densités de déformation sont visibles. Par conséquent, nous affinons actuellement la structure en utilisant des méthodes d’affinage multipolaires.

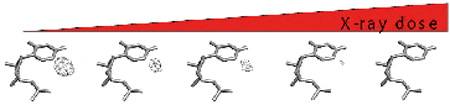
Développement d’une nouvelle approche par étapes : la méthode RIP. Nous avions signalé qu’une des principales causes d’échec de la technique MAD avec les bromo- ou iodo-uridines était due au clivage de l’atome d’halogène induit par les rayons X. En considérant le clivage de ces diffuseurs anomaux comme générant un “natif” à partir d’un cristal “dérivé”, nous avons pu compléter la perte progressive du signal anomal utilisé dans le phasage MAD par ce signal concomitant de type MIR. Nous avons maintenant renforcé la méthode en montrant comment surveiller l’étendue du clivage à tout moment pendant la collecte des données.
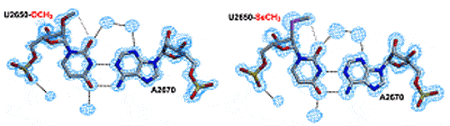
Une alternative pour la mise en phase MAD consiste à utiliser de l’ARN séléné (résistance accrue aux radiations). Cependant, la modification de l’ARN en remplaçant un ou plusieurs groupes 2′-OH par un ou plusieurs groupes 2′-SeCH3 est chimiquement lourde et coûteuse. Nous avons donc développé une stratégie basée sur le criblage préliminaire de la cristallisation de l’ARN modifié en 2′-OCH3, avant le remplacement des groupes 2′-OCH3 par leurs homologues 2′-SeCH3.

Nous avons mis en œuvre des développements méthodologiques majeurs en matière de calorimétrie de titrage isotherme (ITC). Tout d’abord, les ITC qui fournissent des informations cinétiques similaires aux données habituellement obtenues par résonance plasmonique de surface, ainsi que des informations thermodynamiques habituellement récupérées par les ITC. En partenariat avec Software for Science Development, notre stratégie kinITC a été mise en œuvre dans le logiciel de traitement de données AFFINIMETER ITC
Deuxièmement, l’ITC incrémentiel permet une dissection étape par étape des réactions successives dans les machines biomoléculaires. Cette approche a été utilisée avec succès pour découvrir le mécanisme d’action de la transcriptase inverse du VIH-1 et le mécanisme d’inhibition des inhibiteurs de la transcriptase inverse du VIH-1 utilisés en clinique.

Les pinces coulissantes multimériques pour l’ADN (également appelées anneaux β) confèrent une grande processivité aux polymérases réplicatives de l’ADN. Ce sont également des centres moléculaires sur lesquels de nombreuses protéines impliquées dans le métabolisme de l’ADN interagissent en se liant, via une séquence peptidique conservée, dans une poche universellement conservée (figure).
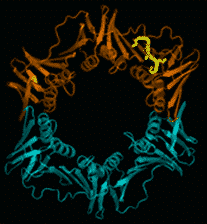
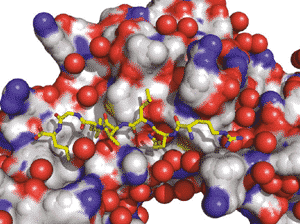
Chez les bactéries, la poche d’interaction est une nouvelle cible potentielle pour le développement de nouveaux composés antibactériens, qui sont nécessaires de toute urgence pour contrôler et surmonter la résistance bactérienne croissante aux antibiotiques.
Nous utilisons une approche basée sur la structure pour concevoir des peptides qui se lient dans la poche d’interaction avec une grande affinité, entrent en compétition avec les polymérases de l’ADN et déclenchent la mort cellulaire. Ce projet est mené grâce à une stratégie multidisciplinaire et intégrée résultant d’une étroite collaboration entre sept laboratoires différents, combinant ainsi la synthèse chimique, la modélisation moléculaire, les analyses structurelles, biophysiques et biochimiques et l’évaluation in vivo de l’efficacité des ligands pour inhiber la croissance bactérienne et les processus infectieux.
Dans le cadre de cette collaboration, notre tâche consiste à produire les anneaux β de différentes origines bactériennes et à analyser l’interaction des peptides nouvellement conçus avec ces anneaux, en utilisant des techniques biophysiques telles que l’ITC, la cristallographie à rayons X et la spectrométrie de masse.
Bibliographie :
- Burnouf D., Olieric V., Wagner J., Fujii S., Reinbolt J., Fuchs R.P. and Dumas P. (2004). Structural and biochemical analysis of sliding clamp/ligand interactions suggest a competition between replicative and translesion DNA polymerases. J Mol Biol, 335(5):1187-1197.
- Wolff P., Olieric V., Briand J.P., Chaloin O., Dejaegere A., Dumas P., Ennifar E., Guichard G., Wagner J., Burnouf D. (2011). Structure-based design of short peptide ligands binding onto the E. coli processivity ring. J Med Chem, 54(13):4627-37.
- Wolff P., Amal I., Oliéric V., Chaloin O., Gygli G., Ennifar E., Lorber B., Guichard G., Wagner J., Dejaegere A. and Burnouf D. (2014). Differential modes of peptide binding onto replicative sliding clamps from various bacterial origins. J Med Chem., 57(18):7565-7576.
- Wolff P., Ennifar E., Guichard G., Burnouf D. and Dumas P. Native ESI mass spectroscopy can help avoiding wrong interpretations from isothermal titration calorimetry in difficult situations. Soumis à American Journal of Mass Spectrometry
Collaborations :
Prof Annick Dejaegere (IGBMC, Illkirch, France), Dr Gilles Guichard (IECB, Pessac, France), Dr Gaetan Mislin (ESBS, Illkirch, France), Dr Vincent Oliéric ( SLS, Villigen, Suisse), Prof Jean Marc Reichardt (IBMC, Strasbourg, France), Dr Jérôme Wagner (ESBS, Illkirch, France).
Financements :
Inserm, AstraZeneca
Brevets :
- Structure cristalline de la protéine du facteur de processivité de l’ADN polymérase et un ligand et cet usage. EP 1639509, 29 mars 2003 – WO2004EP06942 20040625. Burnouf D, Wagner J, Dumas P, Fujii S, Fuchs R, Olieric V.
- Compounds binding to the bacterial beta ring. EP11162733.7, dépôt EPO le 15 avril 2011, délivré le 7 septembre 2012. Burnouf D, Dejaegere A, Guichard G, Oliéric V, Wagner J.
Selection de publications
Simonetti A, Guca E, Bochler A, Kuhn L, Hashem Y. Structural Insights Into the Mammalian Late-Stage Initiation Complexes. Cell Rep (2020), 31 (1):107497. https://www.ncbi.nlm.nih.gov/pubmed/32268096?dopt=Abstract
Brillet K, Martinez-Zapien D, Bec G, Ennifar E, Dock-Bregeon A C, Lebars I. Different views of the dynamic landscape covered by the 5′-hairpin of the 7SK small nuclear RNA. RNA (2020), 26 (9):1184-1197.https://pubmed.ncbi.nlm.nih.gov/32430362/
Auffinger P, Ennifar E, D’Ascenzo L. Deflating the RNA Mg 2+ bubble. Stereochemistry to the rescue! RNA (2020), 27 (3):243-252. https://doi.org/10.1261/rna.076067.120
D’Ascenzo L, Vicens Q, Auffinger P. Identification of receptors for UNCG and GNRA Z-turns and their occurrence in rRNA. Nucleic Acids Res (2018), 46(15):7989-7997. https://www.ncbi.nlm.nih.gov/pubmed/29986118?dopt=Abstract
Bec G, Meyer B, Gerard MA, Steger J, Fauster K, Wolff P, Burnouf B, Micura R, Dumas P, Ennifar E. Thermodynamics of HIV-1 reverse transcriptase in action elucidates the mechanism of action of non-nucleoside inhibitors. J Am Chem Soc (2013);135(26):9743-52. https://pubmed.ncbi.nlm.nih.gov/23742167/
Ennifar E, Aslam MW, Strasser P, Hoffmann G, Dumas P, van Delft FL. Structure-guided discovery of a novel aminoglycoside conjugate targeting HIV-1 RNA viral genomeACS Chem Biol (2013);8(11):2509-17. https://pubmed.ncbi.nlm.nih.gov/24015986/
Burnouf D Y, Ennifar E, Guedich S, Puffer B, Hoffmann G, Bec G, Disdier F, Baltzinger M, Dumas P. kinITC: a new method for obtaining joint thermodynamic and kinetic data by Isothermal Titration Calorimetry. J Am Chem Soc (2012), 134(1):559-565.
http://www.ncbi.nlm.nih.gov/pubmed/22126339
Freisz S, Bec G, Radi M, Wolff P, Crespan E, Angeli L, Dumas P, Maga G, Botta M, Ennifar E. Crystal structure of HIV-1 reverse transcriptase bound to a non-nucleoside inhibitor with a novel mechanism of action. Angew Chem (2010), 49(10):1805-8. https://pubmed.ncbi.nlm.nih.gov/20135654/
Ennifar E, Paillart J C, Bodlenner A, Walter P, Weibel J M, Aubertin A M, Pale P, Dumas P, Marquet R. Targeting the dimerization initiation site of HIV-1 RNA with aminoglycosides: from crystal to cell. Nucleic Acids Research (2006), 34(8):2328-39. https://pubmed.ncbi.nlm.nih.gov/16679451/
Soutiens financiers



 Intranet
Intranet Acces
Acces Contact
Contact
