L’initiation de la traduction des ARNm structurés et son contrôle
Chez les bactéries, l’initiation de la traduction et en particulier la reconnaissance des ARNm par le ribosome sont en grande partie responsables de l’efficacité de la traduction d’un ARNm donné. Les régions 5’ non codantes des ARNm conditionnent le devenir de l’ARNm. Elles contiennent souvent les signaux de reconnaissance pour divers régulateurs (protéines, ARN, métabolites), qui peuvent répondre à des changements de l’environnement (pH, température, concentration des ions, ROS). Les projets de l’équipe se focalisent sur la manière dont les ARNm structurés interagissent avec le ribosome, et sur la caractérisation de nouveaux mécanismes de régulation impliquant la structure de l’ARNm chez Escherichia coli and Staphylococcus aureus.
ARN thermosenseur et la réponse au froid chez Escherichia coli
Collaboration avec AM Guiliodori et C. Gualerzi (Université de Camerino, Italie).
Contact : S. Marzi
La température est un des facteurs clés de l’environnement auxquels les bactéries doivent faire face. Lors d’une chute brutale de la température, la transcription et la traduction sont fortement affectées alors qu’une douzaine de gènes codant pour des protéines chaperones de l’ARN, dont la protéine CspA, sont préférentiellement traduits. Ces protéines aident au repliement des ARN à basse température et/ou modifient les structures inactives. En collaboration avec l’équipe de C. Gualerzi, nous avons montré que la structure de l’ARNm cspA entier (in vitro et in vivo) est différente à basse (15°C) et haute (> 30°C) température. L’ARNm naissant adopte une structure qui est instable et transitoire à haute température, mais qui est stabilisée à basse température (Figure) pour être préférentiellement traduite (à basse température). Cette étude met en lumière un nouveau mécanisme moléculaire où le changement de structure n’aurait lieu qu’au cours de la transcription de l’ARNm à basse température.
Le rôle de la protéine chaperone CspA dans le repliement correct de son propre ARNm pour faciliter sa traduction lors du choc froid est actuellement étudié.
Le mécanisme de piège du ribosome et reconnaissance des ARNm structurés par le ribosome
Collaboration avec B. Klaholz et M. Yusupov (IGBMC, Illkirch).
Contacts : S. Marzi, P. Romby
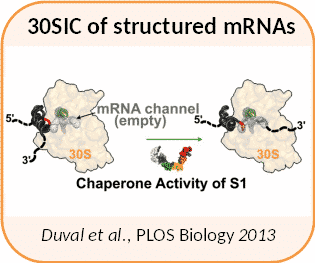
Chez E. coli, de nombreuses protéines fixant l’ARN régulent leur propre synthèse en perturbant la formation du complexe d’initiation de la traduction. C’est le cas de la protéine ribosomique S15 qui reconnait une région de son ARNm qui mime partiellement le site de reconnaissance à l’ARNr. Par des expériences de cryo-microscopie électronique, nous avions montré pour la première fois comment la protéine S15 est capable de piéger l’ARNm sur le ribosome pour former un complexe d’initiation non productif. En stabilisant la formation d’un pseudo-noeud sur la plateforme de la sous-unité 30S, le codon AUG se trouve piégé en dehors du site de décodage pour empêcher la formation de l’interaction ARNt-ARNm. Cette étude a révélé que le ribosome reconnaît les ARNm structurés en deux étapes principales : ancrage de l’ARNm au niveau de la plateforme de la sous-unité 30S suivie d’une étape d’accommodation qui permet de positionner le codon d’initiation dans le site de décodage.
La protéine ribosomique S1 en action sur le ribosome, au carrefour de l’initiation de la traduction et de son contrôle
Collaboration avec E. Masse (Université de Sherbrooke, Canada), B. Luisi (Université de Cambridge, UK) et M. Springer (IBPC, Paris)
Contacts : S. Marzi, I. Caldelari, P. Romby
Nous avons montré que la protéine clé du ribosome qui permet de guider l’ARNm dans le canal de décodage est la protéine S1. Cette protéine qui possède 6 domaines de liaison à l’ARN confère au ribosome des propriétés de « déroulase ». S1 agit de manière différente selon les signaux présents sur l’ARNm, et débobine les structures des ARNm sur le site de la plateforme de la sous-unité 30S pour favoriser l’interaction entre le codon d’initiation et l’anticodon de l’ARNt initiateur. L’activité de « déroulase » de la protéine S1 est portée par ses trois premiers domaines OB. Le premier domaine est multifonctionnel, il est requis pour la fixation au ribosome mais de manière surprenante est aussi essentiel à la fixation de l’ARNm. En revanche, la délétion des domaines 5 et 6 de S1 cause un phénotype froid sensible, et une analyse protéomique différentielle montre que ces deux domaines sont requis pour la régulation de la synthèse de plusieurs protéines.
Ce résultat suggère un rôle clé de S1 dans la régulation de l’expression des gènes chez E. coli que nous sommes actuellement entrain d’exploiter. Parallèlement nous analysons comment les fonctions de S1 ont évolué chez S. aureus. Contrairement à E. coli, la protéine S1 est plus courte et ne se fixe pas au ribosome.
Le ribosome de Staphylococcus aureus et la traduction
Collaboration avec Y. Hashem (UPR 9002), M. Yusupov (IGBMC, Illkirch)
Contacts : Y. Hashem, S. Marzi, I. Caldelari, P. Romby
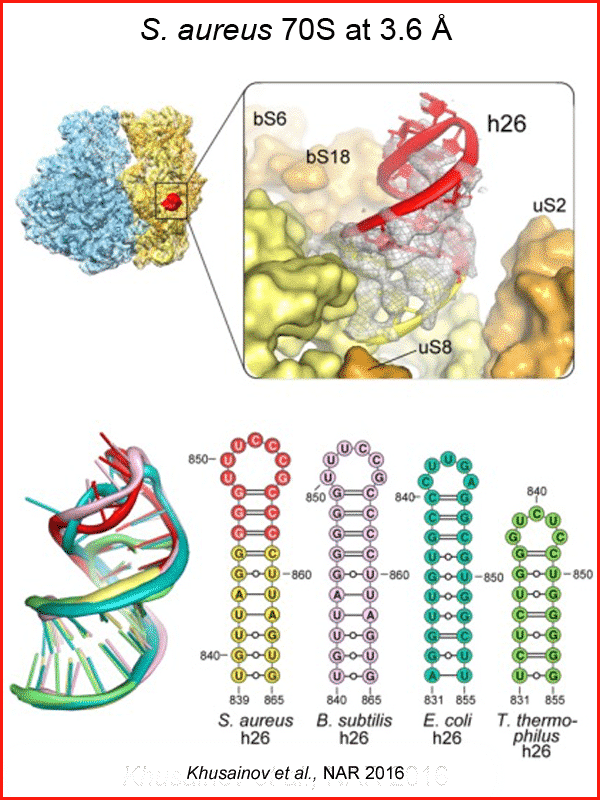
La structure du ribosome de S. aureus a été récemment résolue par cryo-EM à 3.6 Å de résolution révélant quelques particularités au niveau de la protéine bl31 et de certaines hélices dont l’une est localisée sur la plateforme de la sous-unité 30S, site de sortie des ARNm.
Notre objectif est d’obtenir une vision détaillée de la traduction chez S. aureus et son contrôle en utilisant une combinaison d’approches (ribosome profiling, traduction in vitro, structure de complexes régulateurs,…).
 Intranet
Intranet Acces
Acces Contact
Contact