Retour
Nos analyses

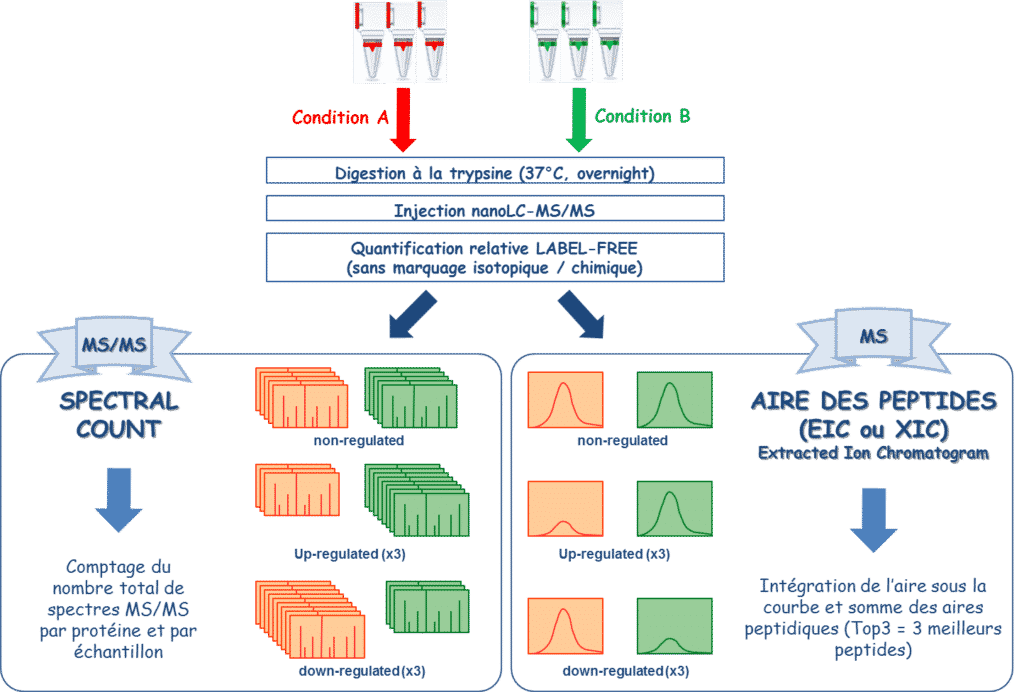
La protéomique est principalement l’analyse des protéines composant un mélange complexe. En spectrométrie de masse “bottom up”, ce sont les peptides issus d’une digestion enzymatique des protéines qui sont analysés. Ces dernières années, la tendance s’est inversée : la digestion à partir de gels SDS-PAGE a laissé la place aux digestions de protéines directement en solution.

Notre plateforme s’est spécialisée dans les analyses de co-immunoprécipitation ou co-IP.
 Intranet
Intranet Acces
Acces Contact
Contact