Des chercheurs du CNRS et de l’Université de Strasbourg ont montré que la protéine NSP1 du virus SARS-CoV-2 provoque des coupures dans les ARNm pendant leur traduction. Ces résultats sont publiés dans la revue Nucleic Acids Reseach le 30/08/2023.
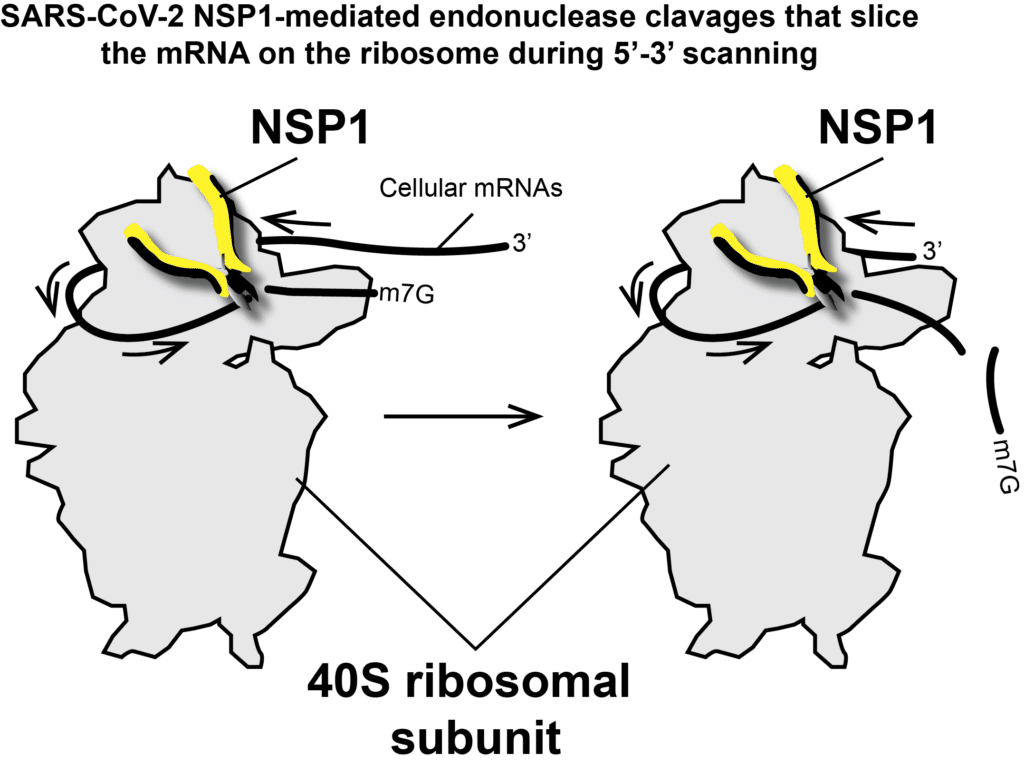
Dans un article publié dans Nucleic Acids Research, des chercheurs du laboratoire Architecture et Réactivité de l’ARN (CNRS, IBMC Strasbourg) ont décortiqué les mécanismes moléculaires qui permettent au virus SARS-CoV-2 d’éteindre la traduction cellulaire pour favoriser la traduction du génome viral par les ribosomes de la cellule infectée. Lors des étapes précoces de l’infection, la protéine NSP1 est l’une des premières protéines virales produites. Cette protéine NSP1 va se fixer immédiatement sur le ribosome de la cellule hôte. Les chercheurs ont montré que la protéine NSP1 sur le ribosome va induire des coupures espacées régulièrement dans les ARN messagers de la cellule au fur et à mesure que le ribosome avance sur les ARN messagers. Ainsi, tous les ARN messagers qui sont traduits seront dégradés en raison de la présence de la protéine NSP1. En revanche, les ARN du virus SARS-CoV-2 échappent à ces dégradations et sont traduits normalement. Les chercheurs ont montré que les ARN du virus sont immunisés contre ces dégradations guidées par la protéine NSP1. En effet, le virus possède dans ses ARN messagers, une petite structure en épingle à cheveux appelée SL1 qui lui permet de protéger ses ARN contre les dégradations sur le ribosome en agissant sur NSP1. Le virus possède en quelque sorte une clé qui lui autorise la poursuite de la traduction virale alors que la traduction cellulaire s’éteint progressivement. Cette découverte est extrêmement importante puisqu’elle ouvre la voie à de nouvelles approches thérapeutiques antivirales visant à bloquer le cycle cellulaire du virus. En effet, le rôle primordial de SL1 dans le processus infectieux fait de cet élément du virus une cible de choix pour le développement de nouvelles molécules inhibitrices. De telles molécules seraient capables de bloquer spécifiquement l’action de SL1 permettant ainsi d’éteindre la traduction du virus. En résumé, le fait que SL1 soit indispensable à la traduction virale suggère que cette structure serait un véritable ‘talon d’Achille’ du virus. Ces travaux ont été financés par la Fondation pour la Recherche Médicale (projet coronaIRES) dans le cadre d’un appel d’offre flash initié par l’Agence National pour la Recherche.

En savoir plus :
SARS-CoV-2 NSP1 induces mRNA cleavages on the ribosome
Yann TARDIVAT, Piotr SOSNOWSKI, Antonin TIDU, Eric WESTHOF, Gilbert ERIANI, Franck MARTIN*
Publié dans Nucleic Acids Research
*corresponding
Contact chercheur :
Franck MARTIN, f.martin@ibmc-cnrs.unistra.fr
Architecture et réactivité de l’ARN
UPR9002 CNRS
Institut de Biologie Moléculaire et Cellulaire
2 allée Konrad Roentgen 67084 Strasbourg cedex
 Intranet
Intranet Acces
Acces Contact
Contact