Répertoire méthodologique
A propos de l’ITC Calorimétrie
La calorimétrie par titrage isotherme (ITC) est l’étalon-or pour la mesure des interactions biomoléculaires. L’ITC détermine simultanément tous les paramètres de liaison (n, K, ΔH et ΔS) dans une seule expérience – des informations qui ne peuvent être obtenues par aucune autre méthode.
Lorsque les substances se lient, de la chaleur est soit générée, soit absorbée. L’ITC est une technique thermodynamique qui mesure directement la chaleur libérée ou absorbée lors d’un événement de liaison biomoléculaire. La mesure de cette chaleur permet de déterminer avec précision les constantes de liaison (KB), la stoechiométrie de la réaction (n), l’enthalpie (ΔH) et l’entropie (ΔS), fournissant ainsi un profil thermodynamique complet de l’interaction moléculaire en une seule expérience. Parce que l’ITC va au-delà des affinités de liaison et peut élucider le mécanisme de l’interaction moléculaire, elle est devenue la méthode de choix pour caractériser les interactions biomoléculaires.
Les applications comprennent :
- La caractérisation des interactions moléculaires de petites molécules, de protéines, d’anticorps, d’acides nucléiques, de lipides et d’autres biomolécules.
- L’optimisation des pistes.
- La cinétique des enzymes.
- L’évaluation de l’effet des changements de structure moléculaire sur les mécanismes de liaison.
- L’évaluation de l’activité biologique.
Études sur la liaison
Tous les processus cellulaires nécessitent une liaison spécifique et une reconnaissance moléculaire entre les biomolécules. La connaissance de ces interactions est importante pour comprendre comment les protéines, les acides nucléiques et les lipides fonctionnent dans les systèmes biologiques. Des progrès rapides ont été réalisés en biologie structurelle et en reliant la structure à la fonction et au mécanisme biochimiques. Cependant, la connaissance de la structure seule ne permet pas de prédire avec précision la fonction et l’activité biologique. La caractérisation complète de toute interaction de liaison nécessite une quantification de l’affinité, du nombre de sites de liaison et de la thermodynamique.
Les données thermodynamiques, en particulier l’enthalpie (ΔH) et l’entropie (ΔS), révèlent les forces qui régissent la formation et le mécanisme d’action complexes. La thermodynamique fournit des informations sur les changements de conformation, la liaison hydrogène, les interactions hydrophobes et les interactions charge-charge. Ces informations peuvent être utilisées pour décrire la fonction et le mécanisme au niveau moléculaire.
Études de liaison avec la calorimétrie par titrage isotherme (ITC)
La calorimétrie par titrage isotherme (ITC) est un outil analytique puissant qui mesure l’affinité de liaison et la thermodynamique entre deux biomolécules quelconques. L’ITC est considérée comme le test “étalon-or” pour la liaison, et présente de nombreux avantages :
- Essai universel – mesure directement le changement de chaleur associé à la liaison
- Sans label – utilise des matériaux indigènes
- Une véritable technique en solution
- Nécessite un développement minimal du test
- Ne présente aucune limitation de poids moléculaire
- Non-optique
- Polyvalent, il peut être utilisé avec n’importe quelle biomolécule – protéines, acides nucléiques, médicaments à petites molécules, lipides, etc.
- Peut être utilisé avec un large éventail de tampons biologiques, de forces ioniques, de pH
Dans une seule expérience de l’ITC, on peut déterminer :
- L’affinité de liaison – Kd
- Mesurer directement de sub-millimolaire à nanomolaire
- Peut étendre la gamme d’affinité au picomolaire en utilisant la méthode compétitive de l’ITC
- Le nombre de sites de liaison
- Des sites de liaison multiples et différents
- L’enthalpie (ΔH) et l’entropie (ΔS) de la liaison
L’ITC est essentiel dans l’étude des relations entre structure et activité des sondes multiples (SAR) car il peut détecter des contributions que les méthodes par affinité seulement peuvent manquer. Par exemple, l’affinité mesurée par ces méthodes peut être similaire pour une protéine de type sauvage et mutante se liant à un médicament, mais l’ITC peut révéler des différences dans ΔH et ΔS qui peuvent décrire le mécanisme d’action de la liaison. Ces informations peuvent valider la modélisation in-silico. L’ITC est également couramment utilisé pour valider d’autres tests de liaison.
L’ITC devient un outil important dans la caractérisation des interactions entre les médicaments et les cibles, et peut être utilisé à différents stades de la découverte et du développement des médicaments. L’ITC est polyvalent et peut être utilisé pour caractériser la liaison entre deux molécules quelconques.
Cinétique des enzymes
Les réactions catalytiques enzymatiques sont au cœur de toutes les voies biologiques. Une grande partie de la recherche biochimique est consacrée à la caractérisation de la fonction, de l’activité et de la structure des enzymes, ainsi qu’à la manière dont les enzymes sont inhibées et activées. La caractérisation des enzymes est également essentielle à la découverte de médicaments, car de nombreuses cibles de médicaments sont des enzymes. Il s’agit notamment des kinases et des protéases.
La calorimétrie par titrage isotherme (ITC) est bien établie dans l’étude de l’affinité des interactions moléculaires, et devient maintenant un outil courant dans l’étude de la cinétique des enzymes. La force de cette technique réside dans la nature universelle de l’ITC. Les analyses enzymatiques traditionnelles utilisent une sonde pour surveiller soit l’épuisement du substrat, soit la formation du produit. Ces sondes sont dépendantes du système et doivent être optimisées pour chaque réaction dans des conditions spécifiques. De plus, il peut être nécessaire de modifier le substrat, ce qui pourrait interférer avec la réaction de catalyse. Pour les méthodes optiques, les conditions expérimentales peuvent affecter le système de détection, empêchant des mesures précises. Cela signifie qu’avec les méthodes d’analyse traditionnelles, de nombreuses enzymes ne disposent pas de tests pratiques.
L’ITC utilise la chaleur comme sonde, et puisque chaque réaction génère ou absorbe de la chaleur, il n’est pas nécessaire de développer une longue méthode à chaque fois qu’une nouvelle enzyme est testée. L’ITC mesure directement le changement de chaleur au fur et à mesure de la catalyse, qui est proportionnel à la vitesse de la réaction. Todd et Gomez (2001) ont montré que les Km et kcat des expériences ITC concordent favorablement avec les méthodes traditionnelles de cinétique enzymatique, et peuvent être utilisés avec toutes les classes d’enzymes, y compris celles qui ne sont pas soumises à d’autres méthodes de dosage direct.
L’utilisation de l’ITC pour surveiller la vitesse des réactions enzymatiques est un test non destructif, sensible et direct. Des injections multiples de substrat peuvent être effectuées en une seule expérience, ce qui permet de déterminer le Km et le kcat en une seule expérience d’ITC. Avec l’ITC, il est facile de faire varier les conditions expérimentales telles que le pH et la force ionique, et on peut obtenir une analyse complète de la catalyse et de la cinétique. L’ITC fournit également de précieuses informations sur la thermodynamique des réactions enzymatiques.
Mesure des affinités de liaison étroites avec l’ITC
La gamme des constantes de liaison qui peuvent être directement mesurées avec la calorimétrie de titrage isotherme (ITC) va du millimolaire au nanomolaire. Avec une liaison serrée (KB >109 M-1 ou Kd plus serrée que la nanomolaire), les titrages ITC perdent leur courbure et l’affinité ne peut être calculée avec précision (Figure1, panel A – Wiseman et al, 1989). Une méthode permettant de mesurer les KB supérieurs à 109 M-1 a été mise au point (Sigurskjold, 2000, Velazquez-Campoy et Freire, 2006). Cette méthode nécessite deux ligands : un ligand à liaison étroite (A) et un second ligand (B) qui a une affinité de liaison plus faible et se lie de manière compétitive au même site que le ligand A. Dans la première expérience ITC (figure 1, panel B), le ligand B est titré dans la solution de protéine, et le KB et ΔH sont déterminés. Dans la deuxième expérience ITC (figure 1, panel C), le ligand A est titré dans le complexe protéine-ligand B et le ligand A déplace le ligand B. En utilisant des modèles appropriés d’ajustement de courbe (Sigurskjold, 2000), la constante de liaison du ligand à liaison étroite peut être calculée. Velazquez-Campoy et al (2001) ont utilisé cette méthode pour étudier plusieurs inhibiteurs de la protéase du VIH-1.
FIGURE 1. Titrages ITC représentatifs de ligands compétitifs se liant au VIH pour la détermination du KB du ligand à liaison étroite.



L’ITC (Isothermal Titration Calorimetry) (site web Microcal) est l’étalon-or pour la mesure des interactions biomoléculaires. L’ITC détermine simultanément tous les paramètres de liaison (n, K, ΔH et ΔS) dans une seule expérience – des informations qui ne peuvent être obtenues par aucune autre méthode.
L’ITC200 est le calorimètre de titrage isotherme le plus sensible du marché, mais il est aussi extrêmement fragile. Pour cette raison, je vais m’en occuper et réaliser votre ou vos expériences (mais pas traiter vos données !).
Filtrer ou centrifuger vos échantillons (très important surtout pour éviter toute obstruction de la seringue).
Échantillons requis : 300 µl d’échantillon dans la cellule
70 µl de ligand dans la seringue (jusqu’à 200 µl si possible)
10 ml de solution tampon
L’échantillon et le ligand doivent être strictement dans le même tampon (sels, pH …). Ceci est extrêmement important pour la mesure de l’ITC ! Toute différence générera du bruit et rendra l’interprétation plus difficile.
Pour obtenir une approximation de la concentration nécessaire dans la cellule, il faut estimer le Kd du système et le multiplier par 10. Avec des liants très serrés, la concentration doit se situer dans la partie basse de cette valeur ; pour des liants faibles, la concentration doit se situer dans la partie haute de cette valeur. Multipliez la concentration cellulaire par n et par 10 – 15 pour la concentration de la seringue. Les concentrations minimales et maximales qui sont couramment utilisées dans l’ITC200 sont de 3 µM à 500 µM.
ΔS et ΔG sont déduits des Kd et ΔH observés : ΔG = -RT log (1/Kd) = ΔH – T ΔS
La réaction de liaison doit être soit endothermique soit exothermique (ΔH ≠ 0), sinon aucun signal ne sera observé.
On obtient une détermination optimale du Kd si 1000 > c > 1, où c = nombre de sites x Ka x concentration. Pour une détermination optimale de ΔH, un plateau doit être obtenu au début de l’expérience.
Traitez vos données soit avec votre programme préféré, soit avec Origin 7 installé sur l’ordinateur de l’ITC200 (voir le manuel ci-dessous).
Contactez-nous pour prendre un rendez-vous E.Ennifar@ibmc-cnrs.unistra.fr ou K.Brillet@ibmc-cnrs.unistra.fr et pour réserver un créneau horaire.
kinITC : a new method for obtaining joint thermodynamic and kinetic data by Isothermal Titration Calorimetry
D. Burnouf, E. Ennifar, S. Guedich, B. Puffer, G. Hoffmann, G. Bec, F. Disdier, M. Baltzinger & P. Dumas
JACS (2011) DOI : 10.1021/ja209057d
http://pubs.acs.org/doi/abs/10.1021/ja209057d
- L’utilisation de méthodes biophysiques augmente le succès de l’obtention de structures cristallines ligandées
- Calorimétrie de titrage isotherme de l’ARN
- Applications de la calorimétrie de titrage isotherme en biochimie et biophysique des ARN
- Changements de capacité thermique associés au repliement des acides nucléiques
- Calorimétrie et thermodynamique dans la conception des médicaments
- Ligand se liant à des macromolécules unidimensionnelles semblables à un réseau : Analyse de la théorie de McGhee-von Hippel mise en œuvre dans la calorimétrie par titrage isotherme
- ITC dans l’ère post-génomique. . . ? Inestimable
- Calorimétrie par titrage isotherme pour déterminer les constantes d’association des ligands à haute affinité
- Thermodynamique de la reconnaissance de l’ARN aminogl.-r
Plage de température de fonctionnement : 2 – 80°C
Quotient de sensibilité de titrage : 8 ncal × ml
Temps de réponse : 10 s
Cellule : 200 µl
Seringue d’injection : 40 µl
Plus petite injection : 0,1 µl

L’ITC200 est désormais capable de réaliser une expérience de liaison complète en utilisant une seule injection continue, contrairement à la procédure normale qui nécessite des injections multiples. Dans cette procédure d’injection unique, une seule injection lente et continue de solution de ligand est effectuée à partir de la seringue d’injection dans la solution de macromolécules contenue dans la cellule de l’échantillon. La technique SIM améliore considérablement la vitesse de l’expérience (généralement 6 à 12 minutes par cycle !) et est particulièrement adaptée à la découverte de médicaments et au criblage de cibles. L’analyse des données doit être effectuée avec le module SIM d’Origin Microcal LLC.
Exemple de SIM réalisée sur notre ITC200 (19/01/2009) et comparaison avec une série d’injections multiples équivalente :
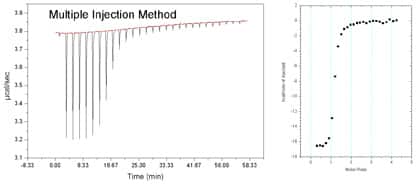

De plus amples informations sur la carte SIM sont disponibles sur le site web de Microcal. Consultez également les documentations disponibles sur ce site.
La dépendance de la température des chaleurs de liaison est un phénomène souvent négligé par les utilisateurs d’ITC. Par conséquent, les études expérimentales peuvent être abandonnées lorsque les chercheurs mesurent l’enthalpie de liaison d’une réaction à une température où la chaleur de liaison est proche de zéro. Dans un tel cas, le rapport signal/bruit est faible, l’isotherme de liaison n’est pas bien défini et les chaleurs de dilution sont comparables aux chaleurs de réaction. Le chercheur peut conclure que l’ITC n’est pas adapté à l’étude de l’interaction d’intérêt. Au contraire, la dépendance de la température de la chaleur de réaction est un outil précieux pour optimiser l’étude de tout événement de liaison.
La chaleur de liaison pour une réaction donnée et le changement d’enthalpie (ΔH) sont généralement dépendants de la température. Lorsque vous modifiez la température d’une expérience, la chaleur d’injection brute et donc ΔH, changent également. La dépendance de la température est due au changement de capacité thermique de l’événement (ΔCp = (ΔH1-ΔH2)/(T1-T2)). La valeur de ΔCp pour les interactions biologiques est presque toujours négative et se situe entre environ 0,3 et 2 kcal/dégré/mole. Si vous collectez une série d’expériences utilisant les mêmes partenaires de liaison à différentes températures et que vous tracez les valeurs ajustées de ΔH en fonction de la température, la pente d’une ligne droite passant par les données est alors la ΔCp. Le ΔCp peut être utilisé pour obtenir des températures de réaction plus élevées et donc de meilleures données. Par exemple, supposons que vous travaillez actuellement à 30°C et que vous ayez obtenu une valeur ajustée pour ΔH = -4 kcal/mol. Comme l’enthalpie de liaison dépend de la température, chaque degré d’augmentation de la température d’une expérience fera augmenter l’enthalpie de liaison de 0,3 à 2 kcal/mole. La chaleur brute augmentera également. Dans ce cas, si vous augmentez la température de l’expérience à 37°C, vous devriez obtenir une enthalpie de liaison comprise entre -6,4 et -18 kcal/mole, des chaleurs brutes plus élevées, un meilleur rapport signal/bruit et une courbe de liaison mieux définie. Pour ce faire, il suffit de modifier la température de votre expérience. Vous n’avez pas besoin de modifier les concentrations de vos réactifs. Vous pouvez également réaliser l’expérience à une température plus basse. Considérez l’exemple ci-dessus. Si la même expérience était réalisée à 5°C, l’enthalpie de liaison et les chaleurs brutes deviendraient plus endothermiques. Si le site ΔCp est à -1 kcal/deg/mol, alors la réduction de la température expérimentale à 5 degrés donnera une enthalpie de liaison de +21 kcal/mol. Les chaleurs d’injection brutes seront également endothermiques et d’une plus grande ampleur que les chaleurs exothermiques initiales observées à 30°C. Comme le site ΔCp est généralement linéaire et négatif, une mesure de l’enthalpie de liaison à deux températures différentes permettra de prédire l’enthalpie de liaison à n’importe quelle température en ajustant les données sur une ligne droite.
La variation de la capacité thermique de liaison n’est pas un concept nouveau. Les utilisateurs de longue date des instruments MicroCal ont utilisé la dépendance de la température de la chaleur de liaison pour optimiser les expériences et obtenir des informations structurelles supplémentaires associées aux réactions de liaison. La plupart des réactions de liaison ont une température à laquelle ΔH = 0. Si vous collectez des données à 5 ou 10 degrés près de cette température, alors les chaleurs seront presque toujours faibles. Si vous modifiez la température de l’expérience, vous augmenterez la chaleur de la réaction et obtiendrez un meilleur signal au bruit. Il est intéressant de noter que de nombreux systèmes ont des valeurs pour ΔH qui passent par zéro entre 20 et 30 degrés.
“N” est le nombre moyen de sites de liaison par mole de protéine dans votre solution, en supposant que :
- tous les sites de liaison sont identiques et indépendants
- vous avez des protéines pures (et un ligand)
- vous avez donné les bonnes concentrations de protéines et de ligands
- toutes vos protéines sont correctement pliées et actives…
C’est rarement le cas dans la pratique ! La détermination de la concentration des protéines (et des ligands) dépend de la précision des méthodes utilisées. Les coefficients d’extinction des protéines, par exemple, sont rarement mieux connus que ±5%, et sont généralement plus mauvais. De mauvaises techniques de mesure, des corrections incorrectes de la ligne de base des UV, des tentatives de conservation du matériel à l’aide de “micro” cuvettes par exemple peuvent entraîner de graves erreurs. Même si toutes vos mesures sont très précises, il se peut que toutes les protéines ne soient pas correctement pliées (une expérience courante avec les protéines recombinantes).
Cas possibles :
N < 1
- la concentration de protéines est plus faible que vous ne le pensez, ou…
- la protéine est impure, ou…
- la protéine (polypeptide) est pure mais pas toute correctement pliée, ou…
- la concentration de ligands est plus élevée que vous ne le pensez, ou…
- un modèle simple de liaison non coopératif est inapproprié, ou…
- tout ce qui précède
N > 1
- votre protéine a de multiples sites de liaison, ou…
- la concentration de ligands est plus faible que vous ne le pensez, ou…
- un simple modèle contraignant non coopératif est inapproprié, ou…
- tout ce qui précède
Aucun signal mesurable, si ΔH est proche de 0 …
Kd hors de portée pour une expérience ITC : si c < 1 la concentration de l’échantillon requis dans la cellule peut être trop élevée, si c > 1000 le déplacement d’un concurrent peut être nécessaire
Aucune information utile ne peut être obtenue à partir d’une …
Autres problèmes :

 Intranet
Intranet Acces
Acces Contact
Contact
















