Flexibilité et évolution génétique du VIH
L’une des principales caractéristiques du virus de l’immunodéficience humaine (VIH) est sa remarquable flexibilité génétique, qui semble être la clé de l’adaptation à la réponse immunitaire de l’hôte et de l’évasion des traitements antiviraux. Cette flexibilité n’est cependant pas illimitée, et la compréhension de ses contraintes au niveau moléculaire est essentielle pour identifier les interactions inter- et intra-géniques qui définissent le réseau épistatique essentiel à la fonctionnalité des protéines et du génome. L’interférence avec ce réseau peut nuire à la réplication virale.
Notre laboratoire s’intéresse à l’étude de la variabilité génétique depuis plusieurs années. Nous avons mis en évidence le rôle central joué par les structures de l’ARN génomique viral dans la génération des formes recombinantes du VIH dans le gène d’enveloppe (figure 1).
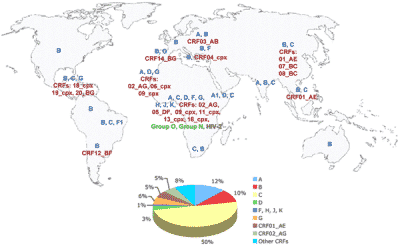
En particulier, nous nous sommes intéressés aux mécanismes de génération et de sélection des formes recombinantes, à partir d’isolats primaires de VIH-1 groupe M. Deux types de VIH ont été identifiés (VIH-1 et VIH-2). Alors que le VIH-2 est limité à des régions géographiques définies en Afrique, le VIH-1 est présent dans le monde entier. Le VIH-1 est subdivisé en quatre groupes M, N, O, et récemment, P. Le groupe M, qui est responsable de la plupart des infections dans les pandémies, est subdivisé en sous-types, appelés par les lettres de A à K (les lettres E et I n’étant pas utilisées). Cette grande diversité est encore amplifiée par l’apparition de recombinaisons entre ces différents types et sous-types. Cela génère ce que l’on appelle des formes recombinantes, qui portent un génome en mosaïque. Ces souches recombinantes pourraient connaître un succès épidémiologique important et sont, par conséquent, définies comme des formes recombinantes circulantes, ou CRF (figure 2).
Certaines de ces formes constituent des souches de VIH prédominantes dans les pandémies. Les mécanismes responsables de la mise en place de ces formes au niveau des pandémies restent largement inconnus.
Le croisement d’isolats naturels de VIH par recombinaison constitue un outil remarquable pour sonder l’existence de réseaux de coévolution et d’interactions épistatiques (figure 3).
En effet, en réunissant, dans un seul cycle infectieux, de grandes portions de génomes d’origine phylogénétique divergente, la recombinaison peut perturber ces réseaux dans la descendance recombinante, en diminuant son aptitude. Les projets développés dans le laboratoire visent à révéler les interactions épistatiques qui limitent la variabilité des protéines virales. Parmi les nombreux gènes du VIH qui nous intéressent, l’enveloppe du VIH est celui qui est le plus étudié. Pour l’enveloppe du VIH, en particulier, il semble y avoir un manque évident d’informations sur les réarrangements spatiaux que la gp120 (la partie extérieure de l’enveloppe) subit pour assurer la réalisation de ses différentes tâches pour une entrée efficace du virus dans la cellule cible. La compréhension des règles qui régissent la dynamique de l’évolution de l’enveloppe du VIH dans les pandémies pourrait être précieuse, non seulement pour comprendre comment le VIH parvient à équilibrer les exigences concurrentes du maintien de la fonctionnalité et de la variation antigénique, mais aussi pour mettre en évidence les aspects vulnérables de la fonctionnalité Env.
Pour aborder ces questions, nous nous appuyons sur l’utilisation d’approches génétiques originales basées sur des essais de culture cellulaire et le laboratoire a plusieurs collaborations internationales bien établies pour développer des approches complémentaires, comme les études phylogénétiques et structurelles.
Liste des collaborateurs :
- David Roberston (Manchester, UK)
- Darren Martin (Cape Town, South Africa)
- Jeff DeStefano (College Park, MD, USA)
- Rafael Sanjuan, Moya Laboratory (Valencia, Spain)
- Cecilia Graziosi, (Lausanne, Switzerland)
- Andrés Finzi, (Boston, MA, USA)
- Vincenzo Di Bartolo, (Paris, France)
- Kevin Weeks (Chapel Hill, NC, USA)
- Eric Arts (Cleveland, OH, USA)
 Intranet
Intranet Acces
Acces Contact
Contact