Évolution des Systèmes d’Initiation de la Traduction chez les Eucaryotes
Chez les eucaryotes, l’initiation de la traduction requiert une pléiade de facteurs d’initiation destinés à recruter la petite sous-unité du ribosome et le tRNAMet initiateur à proximité de l’extrémité 5’ coiffée. Une fois cet assemblage réalisé, un processus de scanning de l’ARNm permet au complexe d’initiation de parcourir l’ARNm jusqu’au premier codon AUG initiateur. Il s’ensuit l’assemblage de la grande sous-unité du ribosome après départ des facteurs d’initiation, puis le démarrage de la traduction.
Afin de gagner en efficacité et pour s’affranchir des facteurs d’initiation certains virus ont appris à contourner l’étape d’initiation classique par un processus baptisé « Internal Ribosome Entry Site » (IRES) qui leur permet de recruter très efficacement les ribosomes à l’intérieur de l’ARNm. Des IRES sont également présents dans certains ARNm cellulaires. Ces éléments entrent en action dans des conditions cellulaires particulières (stress, cycle cellulaire).
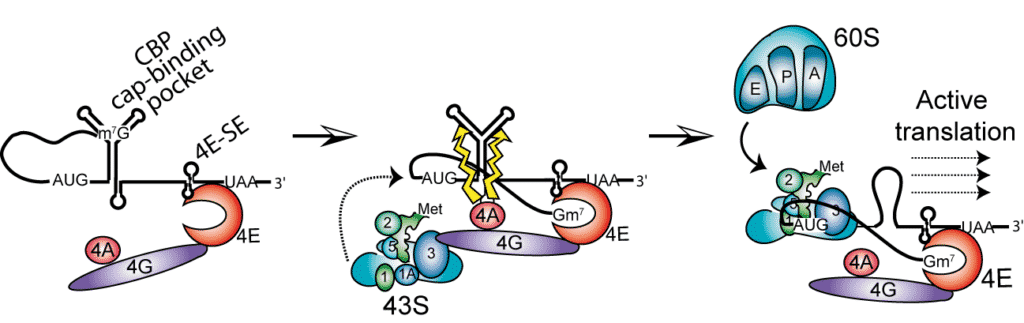
Notre laboratoire s’intéresse aux mécanismes d’initiation de la traduction non conventionnels. Notre système modèle est l’ARNm de l’histone H4 humaine. Cet ARNm est caractérisé par une très grande conservation de séquence, par une très courte région 5’ non traduite et une absence d’extension poly A en 3’, toutes ces caractéristiques semblant exclure l’utilisation d’un mécanisme d’initiation conventionnel utilisant le « scanning » par le ribosome. Nos travaux ont permis de décrire un nouveau type de mécanisme d’initiation appelé « tethering » permettant à l’ARNm de l’histone H4 d’être traduit très efficacement (Martin et al., 2011).
L’ARNm H4 contient dans sa phase codante 2 éléments structuraux critiques pour l’initiation de la traduction. Un des éléments fixe le facteur d’initiation eIF4E sans l’aide de la coiffe. A partir de ce site, la petite sous-unité ribosomale activée 43S est recrutée par les facteurs puis déposée directement sur l’AUG initiateur sans faire appel au scanning de l’ARNm par le ribosome. La seconde structure essentielle de l’ARNm est localisée 19 nucléotides après l’AUG initiateur. Elle se replie en jonction à 3 hélices et séquestre la coiffe tout en facilitant le positionnement du ribosome sur l’AUG initiateur. L’action de l’hélicase eIF4A permet ensuite de libérer le ribosome pour l’étape d’élongation. Ce mécanisme simplifié de « tethering » assure un démarrage immédiat de la traduction, sans avoir recours au « scanning », lent et coûteux en énergie. C’est la première fois qu’un tel processus est démontré pour un ARNm cellulaire.
En savoir plus : « Quand la cellule se prend pour un virus »
En savoir plus : Communication INSB
En savoir plus : Commentaires

 Intranet
Intranet Acces
Acces Contact
Contact